化学位移是核磁共振(NMR)中最重要的物理观测量。它的数据获取相当方便,测量的精确度和可重复性都很高,而且对于生物大分子的结构变化非常敏感,因此成为获取大分子的结构和动态信息的重要数据来源。几十年来,如何准确预测化学位移一直是NMR研究中的一个基础并且关键的问题。一个准确的化学位移预测工具不仅能够简化繁杂的谱峰指认工作,更重要的是,它在生物大分子的结构解析和基于实验数据的结构建模方面有巨大的应用价值。目前对于蛋白质的化学位移预测已经有很多研究,但是对于RNA的化学位移预测却明显滞后,特别是缺乏可靠的针对RNA亚氨基团的预测工具。
清华大学生命科学学院薛毅课题组一直致力于运用以核磁共振为主的实验技术并结合计算方法来研究生物大分子的结构与动态。亚氨基团是对RNA进行核磁测量的重要检测“探针”,但是准确预测其化学位移预测具有较大的难度。在本项研究中,他们发现对于RNA的A型螺旋区,一个亚氨基的化学位移是由其所在的配对以及紧邻的上下两对配对所共同决定的。这样的三对连续碱基对可以简称为碱基对三联体。通过采集几十个发卡RNA样品的化学位移数据,并将这些新数据与核磁公共数据库的已有数据结合,他们得到了一个从碱基对三联体查询亚氨基化学位移的查询表,该表能够用来预测处于碱基对三联体中央的亚氨基的化学位移,并具有很高的预测准确度(图1)。他们还发现不同的碱基对三联体之间化学位移的差异能够通过半经验性模型的计算得到很好的解释。进一步,他们利用半经验性模型计算了部分非经典结构元件的亚氨基化学位移,也达到了相当高的预测准确度。这证明半经验计算模型未来有望能够被用来预测更复杂RNA结构的化学位移。最后,他们还展示了该化学位移预测方法的一个运用实例,即用来确定RNA瞬态的二级结构。他们通过运用15N R1r和1HN CEST核磁弛豫弥散实验,并结合亚氨基化学位移预测方法,成功验证了P5abc RNA瞬态的二级结构,并且证实了之前研究中猜测的G•G碱基错配的存在。
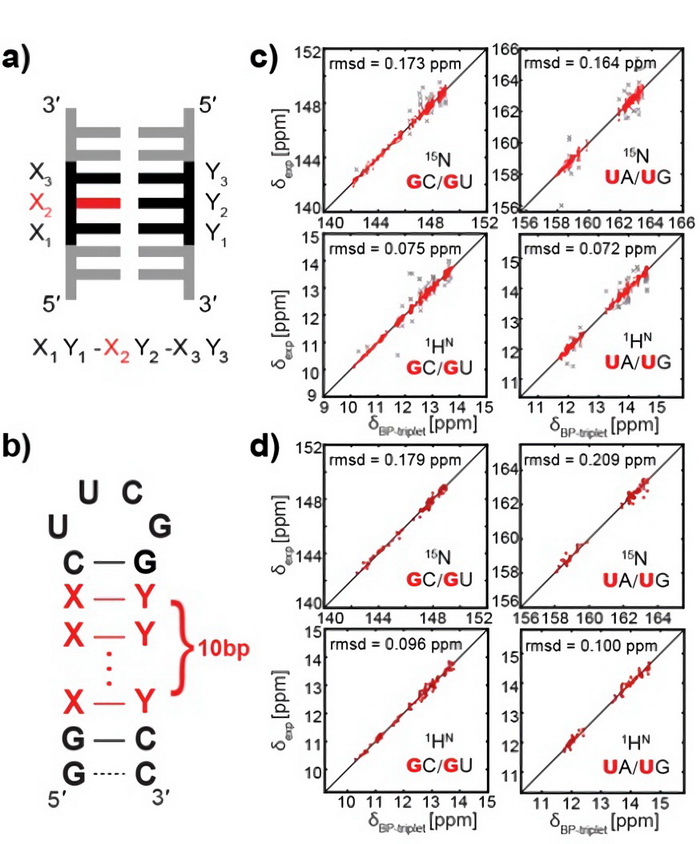
图1. a) “碱基对三联体”的定义;b) 发卡RNA的二级结构;c) 化学位移预测值与实验值的相关性(训练集);d) 化学位移预测值与实验值的相关性(测试集)。
该研究工作2021年3月11日在《自然通讯》(Nature Communications)刊物上发表,题为“RNA化学位移的预测及其在研究RNA瞬态结构上的应用”(Chemical Shift Prediction of RNA Imino Groups: Application toward Characterizing RNA Excited States)。清华大学生命科学学院薛毅研究员为本文通讯作者,生命科学学院已毕业的博士生王艳娇为本文第一作者,生命科学学院博士生韩鸽以及科研助理姜修英参与了部分工作,北京大学药学院宇文泰然研究员在核磁实验的脉冲序列和实验设置上提供了重要帮助。本研究得到了清华-北大生命科学联合中心和北京市结构生物学高精尖创新中心的基金支持。核磁实验数据的采集在清华大学蛋白质研究中心核磁平台完成。
原文链接为:https://www.nature.com/articles/s41467-021-21840-x。
转载链接:http://life.tsinghua.edu.cn/info/1131/2665.htm