大多数生理与病理组织都具有高度的细胞异质性。对异质细胞谱图的系统解读以及关键细胞亚型的精准鉴定对于理解组织发育及其功能、细胞互作微环境、疾病动态发展等生理与病理过程具有重要价值。当前主流的细胞异质性分析以单细胞转录组数据为基础,使用基因表达谱信息定义细胞类群,绘制了大量复杂生理与病理组织的异质性谱图。但是长期以来,这一类异质性分析停留于基因层面,未能达到基因转录本的解析度,几乎完全忽略了RNA可变剪接在塑造生理状态、执行生物学功能及驱动疾病发展过程中的关键作用。
高等真核生物中的RNA可变剪接是重要的转录后调控机制。通过剪接模式的变化,细胞中同一个基因可以生成多个不同的转录本,极大扩展了转录组及蛋白组的复杂性,体现了基因表达及功能的多样性与可塑性。传统的基因表达谱数据仅能反映基因总体表达丰度,无法表征转录后RNA剪接这一重要调控层级。因此,复杂组织的细胞异质性分析有必要补足RNA剪接谱这一关键维度的缺失。
针对这一重要需求,清华大学生命学院杨雪瑞课题组开发了生物信息工具SCASL (single-cell clustering based on alternative splicing landscapes),用于单细胞RNA剪接谱异质性的系统、定量分析,基于剪接谱重新定义细胞亚群。该研究成果于3月9日发表于《自然通讯》(Nature Communications),文章标题为“使用SCASL对单细胞RNA剪接谱的解析提出了具有生理学意义的细胞身份新定义”(Interrogations of single-cell RNA splicing landscapes with SCASL define new cell identities with physiological relevance)。
SCASL方法使用单细胞RNA测序数据,在不依赖于转录组注释的前提下,对不同的可变剪接模式进行定量。以此为基础,SCASL通过kNN插补,重点解决了单细胞RNA剪接数据的低覆盖度与高稀疏性问题,最后使用谱聚类方法对细胞亚型进行了无监督分类,绘制单细胞RNA剪接异质性图谱。测试结果显示,SCASL在对稀疏数据的插补准确性、细胞聚类稳定性、低频细胞类群鉴定、随机噪音容忍度等方面均表现出色,这也体现出单细胞剪接谱所包含信息的高冗余度。更重要的是,基于相同的单细胞RNA-seq数据,SCASL利用剪接谱信息对细胞身份的定义与仅仅基于基因表达谱的细胞聚类结果有显著差异。
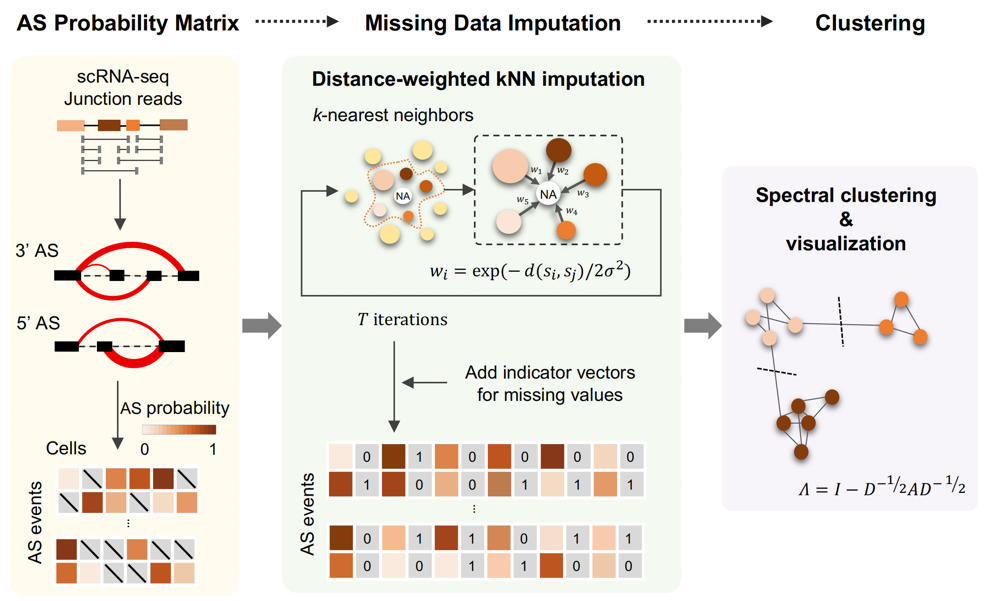
图1. SCASL分析流程概念示意图
进一步,作者使用了七套已发表的单细胞RNA-seq数据,对SCASL剪接谱异质性分析结果的生物学意义进行了深入挖掘和讨论。
首先,通过对三阴性乳腺癌细胞剪接谱异质性的分析,SCASL明确定义了正常上皮细胞的癌前发展状态、早期肿瘤细胞、恶性肿瘤细胞及微转移肿瘤细胞簇。事实上,由于肿瘤细胞高度的异质性,特别是基因表达谱的无序扰动,此前对肿瘤单细胞基因表达谱的聚类通常不够稳定,难以明确定义细胞亚型及其边界。SCASL的分析结果表明RNA剪接谱对于定义细胞亚型,特别是过渡态细胞,具有独特优势,表明RNA可变剪接在驱动肿瘤发生发展中潜在的重要作用。
在对正常组织发育动态过程的解读中,SCASL也表现出独特的优势。例如,SCASL重现了小鼠胚胎肝脏中肝母细胞发育为肝细胞和胆管细胞的两个谱系,明确定义了分化谱系中的时序过渡态细胞。更有趣的是,SCASL基于剪接谱的分析揭示了一个特殊的细胞簇,它此前被定义为胆管细胞,但其剪接谱位于肝细胞和胆管细胞两个发育谱系之间,且同时表现出两种细胞的功能特征,表明其可能代表了肝脏发育过程中两个细胞分化路径之间的交流。
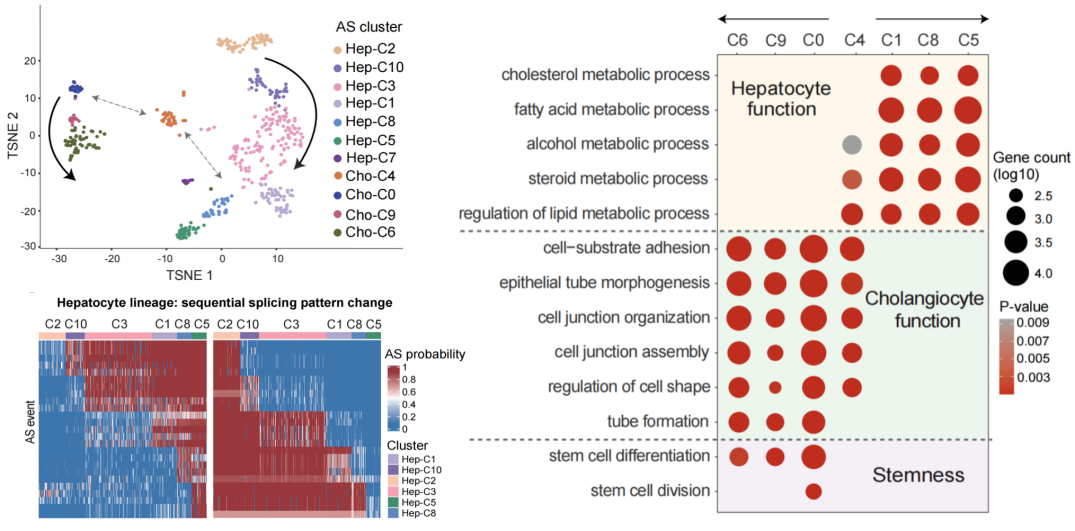
图2. SCASL重现小鼠胚胎肝细胞发育谱系(左),发现肝细胞和胆管细胞两个谱系间的中间态细胞(右)。
最后,在对肿瘤免疫微环境的分析中,SCASL完成了对肿瘤相关T细胞亚型的精细鉴定,包括肿瘤浸润的耗竭型、激活型CD8 T细胞、不同功能活性的CD4 T细胞等。以上新的细胞身份定义为理解免疫细胞浸润、激活与耗竭、细胞互作调控等重要过程提供了新的线索,特别是重要基因的RNA可变剪接对肿瘤免疫微环境的塑造。
总之,本项工作为复杂组织中细胞异质性的解读提出了新的视角与方法。通过这一技术的应用,本研究深入分析了多个重要的生物学过程,提出一系列新观察、新线索与新发现。清华大学生命学院2019级博士生向贤珂为论文第一作者,杨雪瑞副教授为论文通讯作者。北京大学生物医学前沿创新中心张泽民教授及团队成员何尧博士为本研究提供了数据、工具等方面的重要贡献。本工作得到国家重点研发计划、国家自然科学基金委、国家高层次人才特殊支持计划、清华大学的资助。国家蛋白质科学研究(北京)设施(清华大学蛋白质研究技术中心)下属生物计算平台、基因测序与分析平台为本研究提供了大力支持。
论文链接:https://www.nature.com/articles/s41467-024-46480-9
转载链接:https://life.tsinghua.edu.cn/info/1131/5616.htm