作者:陈亚兰
细胞划痕实验是一种在体外研究细胞迁移能力的常用方法。该方法常用于研究肿瘤细胞的侵袭转移能力和细胞基质对于细胞迁移的影响。具体操作为在密集生长的贴壁细胞中间划一道直线,冲洗划痕区后,将细胞放入能够长时间培养观察的显微成像装置,每隔一定时间进行拍照记录,获得多个时间点的图片序列。划痕实验在一定程度上模拟体内细胞迁移的过程。其中主要是以划痕面积或划痕内细胞个数变化来反映迁移的速度。Cell-IQ无标记活细胞成像系统作为一种无损长时程相差成像系统非常适用于细胞划痕实验,其配套的Cell-IQ Analyser软件可一站式设定分析程序并对划痕实验进行批量处理。今天小编就基于Analyser软件来详细介绍下如何进行批量划痕实验分析。
Cell-IQ Analyser软件的分析思路为先建立通用的分析方法,再以此为基础进行批量分析。接下来我们进入分析模块。
建立划痕分析protocol
·Open images
打开软件Cell-IQ Analyser,并打开需进行分析的序列图片File->Open Grids and Wells。主工具栏中选择Tools->Scratch wound measurement 进入划痕分析界面。
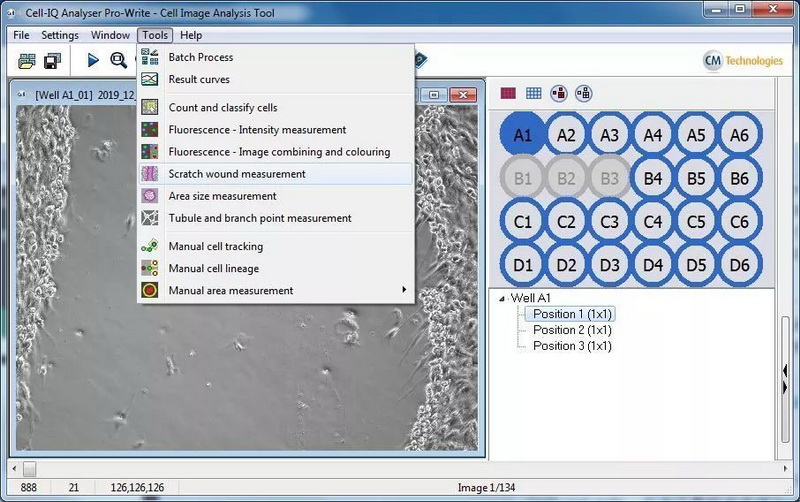
Cell-IQ Analyser分析界面
主窗口为已打开的细胞划痕实验图片,右侧为该实验样品拍摄信息,实验使用24孔板,实际拍摄21个孔。Tools中具有多种分析工具,包括细胞计数、荧光强度分析、多通道图片叠加、细胞追踪和细胞谱系追踪等分析方法。
·Select analysis parameters
分析界面:主窗口显示已打开图片,左侧为分析步骤,当分析时主窗口实时显示分析结果。
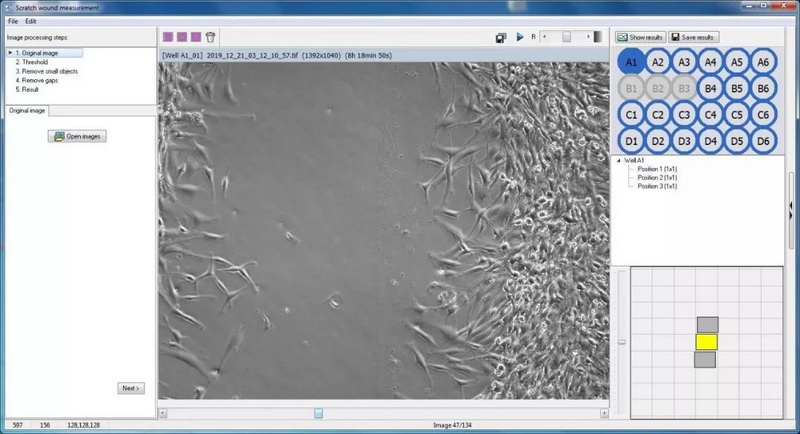
Cell-IQ Analyser 划痕分析界面
主窗口为已打开的细胞划痕实验图片,可拉动下侧进度条观察不同时刻样品信息,右侧为该实验样品拍摄信息,左侧为图片分析进程与参数设置窗口。
Original image 确认当前图片,如需更改可重新打开图片,确认后进入Next。
Threshold 设置分析阈值,调整阈值确保红色蒙版覆盖住所有细胞并且无背景被选入,可试着移动时间序列确定当前阈值的适用性,阈值越低,选中范围越多,Next。
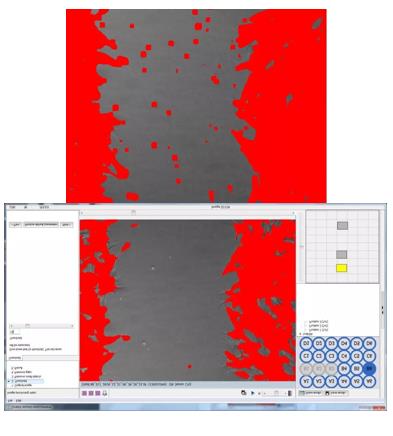
Cell-IQ Analyser 划痕分析界面--Threshold
红色区域为设置阈值包含范围,为细胞所在位置。上下图阈值分别为3,15。仪器有默认推荐值,可选择或自行设定更优参数。
Remove small objects 可去除背景中被多选的点,同时可通过调节erosion和dilation参数来控制去除的点数。erosion与dilation数值越高,被移除的点越多,试着移动时间序列优化参数,Next。
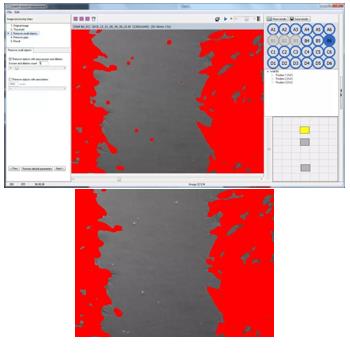
Cell-IQ Analyser 划痕分析界面--Remove small objects
上图划痕中部具有多个红色小点,下图为已去除图片,去除部分信号明显可见是杂信号。数值上图为5,下图为23。仪器同样有默认推荐值。
Remove gaps 连接断面,移除小的缺口(红色部分进行填充),数值越大,细胞间连接越紧密,Next。
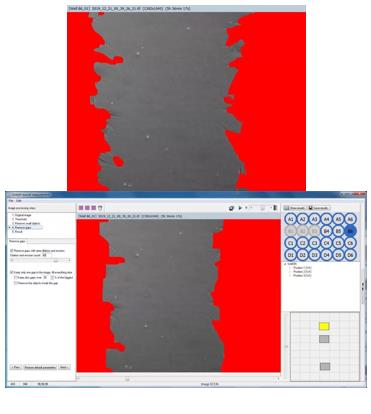
Cell-IQ Analyser 划痕分析界面--Remove gaps
红色区域为设置阈值包含范围,上下图阈值分别为3,80。仪器有默认推荐值。
Result 显示结果,可使用不同的预览呈现方法
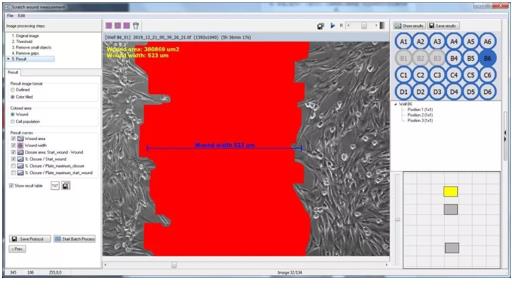
Cell-IQ Analyser 划痕分析界面--Result
红色区域为划痕区域(非细胞区域),黄色字体显示当前划痕面积为380869um2,划痕宽度为523um。左侧可选择结果曲线呈现形式。
Show Result 可以显示分析曲线

预览划痕变化的多种方案

结果分析曲线可呈现的多种方案
图上分别为划痕的水平宽度(wound width)、划痕愈合面积(Closure area)、愈合面积与最初划痕面积的比值(Closure:Start_Wound)随时间变化曲线
Save Protocol
保存分析参数,方便后续批量处理调用。
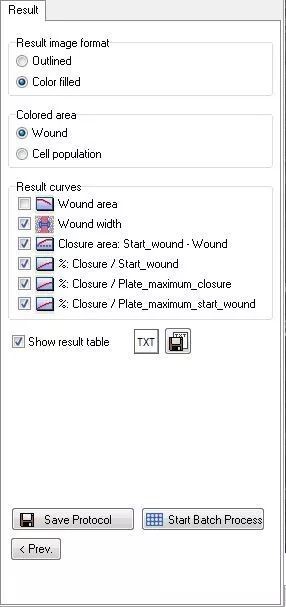
保存protocol与进行批量分析
批量分析
Start Batch Process
进入批量处理模块,先设置数据导入文件位置与数据分析结果保存位置。Next。
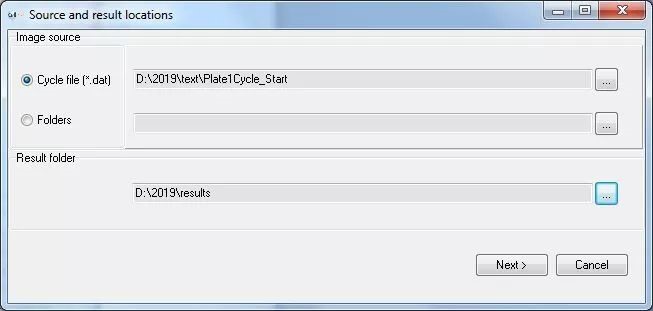
数据打开与保存设置
Image series selection
进入批量分析选择模块,主窗口显示当前选中位置图片,右侧显示样品拍摄信息,可勾选全部图片(红色方格)或勾选部分(圆形红色方格),确定需要进行分析的数据后,Next。
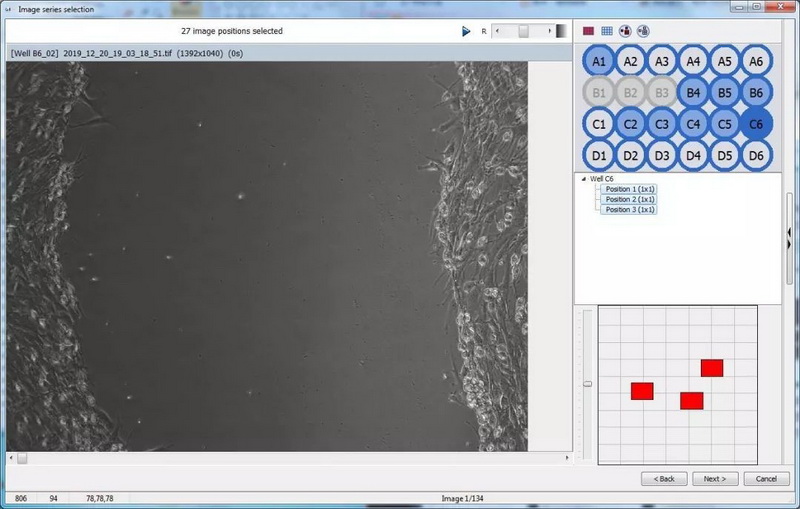
Image series selection界面
图中右侧淡蓝色区域为拍摄选中孔位,中间填充色为淡蓝色为将进行分析的孔位,红色部分为孔内选中的点。
Image file selection
图片分析选择,可以选择部分时刻或循环位置分析数据,结束数据导入,Next进入分析参数设置步骤。
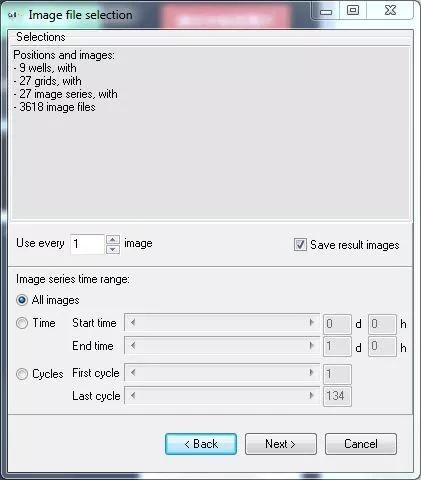
Image file selection窗口
Scratch wound measurement
进入划痕分析参数设置界面,可直接调用已保存的protocol,选择START processing进行批量分析,弹出窗口显示分析进程,同时保存分析结果。
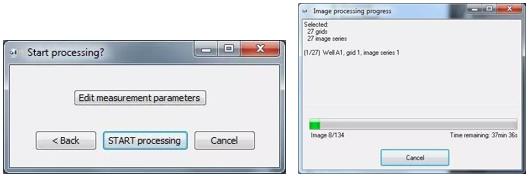
分析进程
start processing 可进行开始分析,也可edit重新设置分析参数进行划痕分析。Image processing process显示正在分析的数据及分析剩余时间。
Analysis results
选择不同实验组别查看实验分析结果,并保存分析数据,并确认数据结果展示方法。
查看分析结果并可导出结果动图
Analysis results界面
选择不同展示方法,并可直接导出分析结果
划痕实验Video
Tips
1. 样品通常需要一段时间稳定。如初始采集图片因温度变化或样品移动导致图片产生偏移,则应舍弃或者进行偏移纠正。偏移可使用FIJI中StackReg或Correct 3D drift等插件进行校正。校正完成后可再进行划痕分析。
2. 细胞划痕实验结果分析还有多种软件可以实现。ImageJ中可通过组合Find Edges、Analyze、measurement进行测量划痕面积分析划痕变化情况,或利用MiToBo等软件分析划痕。
3. 无标记活细胞动态分析系统(Cell-IQ)是一个集培养管理、观察记录和分析于一身的一体式无标记活细胞 (胚胎)动态观察与分析平台。其集成的培养系统可以实现CO2流速、温度的高精度控制,同时采用对细胞低损伤的LED光源,从而使细胞在最优的生长环境下维持一个健康稳定的状态以进行长期的培养观察和记录。
资料参考:
https://www.le.ac.uk/biochem/microscopy/pdf/Wound%20healing%20assay.pdf
https://biii.eu/wound-healing-assay-analysis-imagej
http://dev.mri.cnrs.fr/projects/imagej-macros/wiki/Wound_Healing_Tool
http://mitobo.informatik.uni-halle.de/index.php/Main_Page
本文章版权归清华大学蛋白质研究技术中心细胞影像平台所有