作者:徐宁
蛋白质相互作用研究的重要性不言而喻,常用的生化研究方法包括GST-pull down、ITC、MST、SPR、BIACORE等方法,这些方法是从分子层面给出蛋白互作的一些结合信息。复合物高分辨率晶体结构是从原子层面给出蛋白互作的结合信息。在此我们介绍一种氨基酸残基层面上的确定相互作用的方法:1H-15N HSQC核磁二维谱。
1H-15N HSQC谱采集:如图一所示,纵坐标是15N的化学位移,横坐标是1H的化学位移,之后通过采集HNCO、HNCACO、HNCA、HNCOCA、CBCACONH和HNCACB三维谱,我们可以归属出每一个氨基酸残基对应的谱峰。在加入相互作用的目的物之后,氨基酸残基发生相互作用,导致谱发生变化,如图一(右)所示。
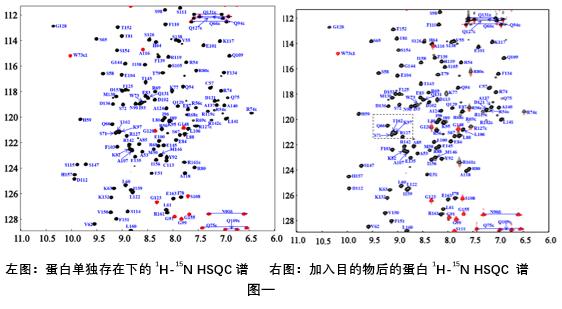
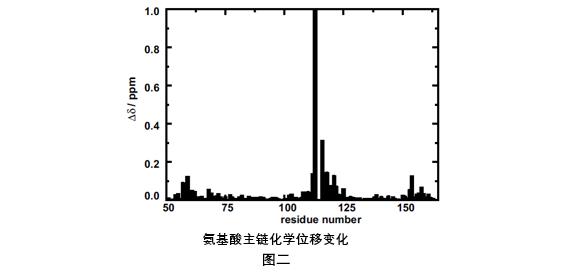
通过比对,我们发现有些位置的氨基酸残基发生明显的偏移,之后我们量化每一个氨基酸残基的化学位移变化,可以很容易判断出哪些氨基酸残基发生了相互作用以及作用最明显的氨基酸残基,如图二所示。
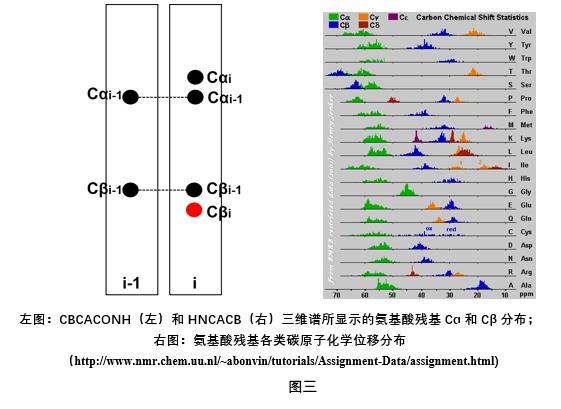
主链归属:1H-15N HSQC中化学位移的归属依赖的是上述六个三维谱图,在实际操作中,为了减少采集时间和节约实验样品,我们有时候只采集CBCACONH和HNCACB。CBCACONH所显示的是前一个氨基酸残基的Cɑ和Cβ信号(Cɑi-1和Cβi-1),HNCACB显示的是自身氨基酸的Cɑ和Cβ(Cɑi和Cβi)以及前一个氨基酸残基的Cɑ和Cβ 信号(Cɑi-1和Cβi-1)(图三)。HNCACB三维谱采集的Cβi 相位信号为负,与其它碳原子相位信号刚好相差180°,在归属中很容易辨认出来, 再结合图三(右)所示的氨基酸碳原子的化学位移分布,我们可以完成大部分的主链化学位移归属。
1H-15N HSQC原理介绍:1H-15N HSQC谱,全称为1H-15N 异核单量子相干谱(1H-15N Heteronuclear Single Quantum Coherence spectroscopy),是生物核磁研究中最基础也是最常用的一个二维谱。图四所示是1H-15N HSQC核磁脉冲序列:
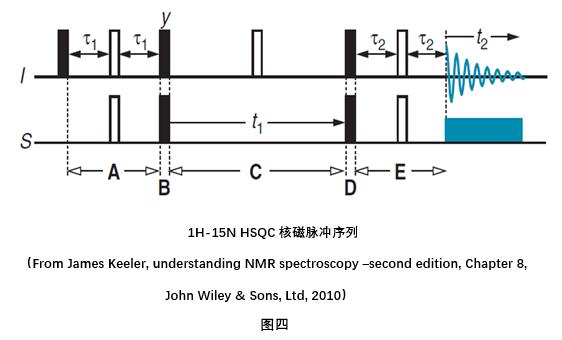
在图四中,自旋I代表1H原子核,自旋S代表15N原子核,该脉冲程序主要包括三个过程:
(1)自旋I的平衡磁化向量转移至自旋S。
在脉冲程序的起始,自旋I处于平衡态Iz,在经过第一个90度硬脉冲后,自旋I的核磁向量被打至xy平面:
紧接着进入自旋回波阶段(spin echo,即图示A阶段),在此期间两核的耦合进行演化:
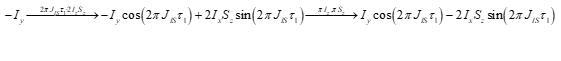
随后的两个90度脉冲(即图示B阶段)将反相项(anti-phase term)转移至自旋S,这也是我们所感兴趣的项,与此同时同相项(in-phase term)不受脉冲影响:
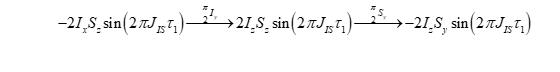
这个过程与一种通过极化转移以增强不敏感核的实验脉冲序列,即INEPT脉冲序列的前半部分相同,因此HSQC脉冲序列的这个初始部分通常也称为INEPT 转移。
(2)t1时段内自旋S的磁化向量的演化。
图示C阶段为t1时段内的演化,在此期间,由于自旋I的频道中心放置了一个180度脉冲,从而形成了一个自旋回波,使得耦合演化重新聚焦,因此此时段内磁化向量的演化等同于自旋S的化学位移偏移(offset)的演化:
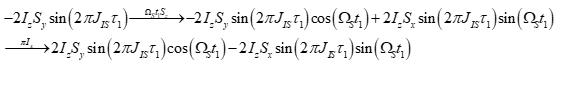
(3)自旋S的磁化向量转移回自旋I以进行观测。
图示D阶段是同时作用在两个核上的90度脉冲,在此作用下上一时段获得的两项分别演变成了自旋I上的反相磁化向量和不可观测的多量子相干项(multiple-quantum coherence):
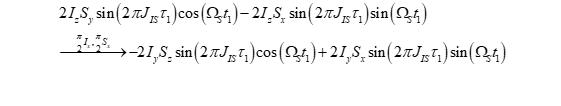
为了去耦合使得反相磁化向量变成同相磁化向量,图示E阶段,即在数据采集之前引入了另一个自旋回波,与A阶段相似,此时段内进行了耦合演化而没有进行化学位移偏移的演化:
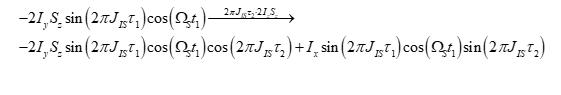
如果现在观测信号,同时将宽带去耦合应用在自旋S上,将会使得在t2时间里磁化向量带上了自旋I的频率标签ΩI。在实验参数设置上,一般将τ1和τ2的长度优化为1/(4JIS),最后对于每个氨基酸的NH基团我们在谱图上将会看到的一个单峰,其坐标对应着N-H这两个耦合自旋的化学位移。
结论:通过以上描述,我们可以采集并归属蛋白质结合前后的1H-15N HSQC谱,通过计算,可以得出参与相互作用的界面以及关键氨基酸,这种方法可以在氨基酸残基层面上为常用生化方法(分子层面)到复合物高分辨率三维晶体结构(原子层面)搭建了一个桥梁。该方法从样品制备到分析出结果,大约需要两周左右的时间,可以为弱相互作用以及无法结晶的复合物研究提供一种强有力的工具。
本文章版权归清华大学蛋白质研究技术中心核磁技术平台所有