作者:李亚妃
COVID-19疫情爆发以来,对SARS-CoV-2的结构,入侵细胞的过程,抗体,抑制剂药物和疫苗的研究进展飞速。分子间相互作用技术可以直接测定分子间的平衡解离常数等,在病毒入侵机理研究,抗体开发,小分子药物筛选和疫苗研发中都能发挥重要作用。
分子间相互作用技术中根据平衡解离常数KD的算法可以分为两大类:平衡法(稳态法)和动力学法。平衡法是在不同配体浓度下定量分析反应平衡时的复合物信息来计算KD值,动力学法则通过测定分子间的结合速率和解离速率,以此计算KD值。蛋白质制备与鉴定平台现有生物膜干涉分子间相互作用仪BLI,表面等离子共振分子间相互作用仪SPR,等温滴定微量热仪ITC,微量热泳动仪MST,分析性超速离心机AUC等多种分子间相互作用分析设备。BLI和SPR分别根据配体分子结合前后发生的光干涉和表面等离子体共振的变化,可实时测定结合和解离过程,并且根据分子结合模式,选择动力学或动力学+稳态的计算方法,同时得到结合速率,解离速率和KD值。ITC是测定分子间互作过程中的热变化;MST是基于生物分子因温度梯度产生泳动的原理检测配体和复合物的热泳动行为的差异;AUC利用紫外/干涉光检测器实时监测样品在超速离心过程中的沉降行为,从而得到样品中各组分的沉降系数,大小,形状等信息,区分复合物和配体。以上仪器均面向校内外用户提供开放测试服务。
病毒入侵机制
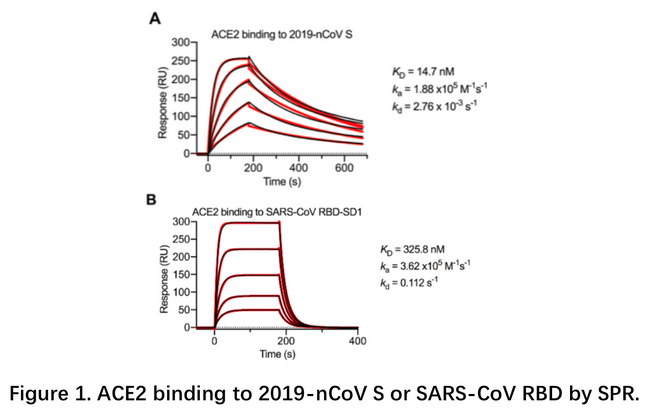
1月22日武汉病毒所石正丽团队在bioRxiv上发文,通过细胞实验证明SARS-CoV-2在细胞表面的受体也是ACE21。后续多个团队先后解析了SARS-CoV-2的S蛋白,受体ACE2,及复合物的结构。其中2月19日德州大学奥斯汀分校和美国国立卫生研究院NIH在Science在线发表了S蛋白的全长三聚体结构,同时分别用SPR和BLI测定了S蛋白全长或RBD与ACE2的结合解离过程。相比SARS-CoV,SARS-CoV-2的S蛋白与ACE2的亲和力高约10倍。借助于动力学分析,发现SARS-CoV-2的解离速率为10-3s-1,而SARS-CoV为10-2 s-1,这可能是SARS-CoV-2传播更快的一个原因。另外文章利用BLI鉴定几种SARS-CoV的抗体并不能结合SARS-CoV-2的S蛋白2。
抗体开发
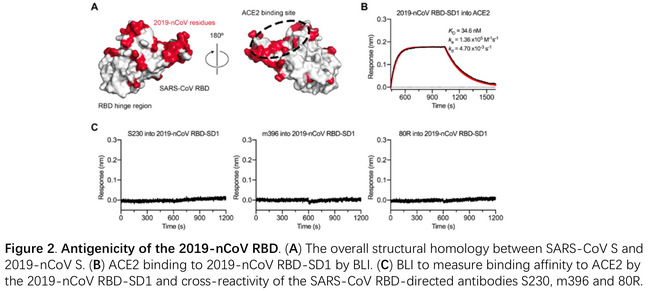
1月28日复旦大学应天雷团队等合作发文,首次发现SARS-CoV单抗CR3022可结合SARS-CoV-2的S蛋白的RBD3。团队利用BLI首先测定了RBD与ACE2的结合解离,得到KD为10-8M,结合速率105M-1s-1 解离速率10-3 s-1。然后测定了几种SARS-CoV单抗与RBD的结合解离,从中发现CR3022的KD达到10-9M,这一结果与ELISA结果相互印证。文章利用BLI进一步测定了CR3022 ACE2 RBD三者的竞争性结合关系。通过固定RBD,依次结合ACE2和CR3022,发现并没有明显的竞争性结合,说明CR3022与ACE2结合RBD的表位并不完全一致。利用BLI测定竞争性结合的方法可参阅https://mp.weixin.qq.com/s/cOIxeAprE8KWqHOgbAAIGw。

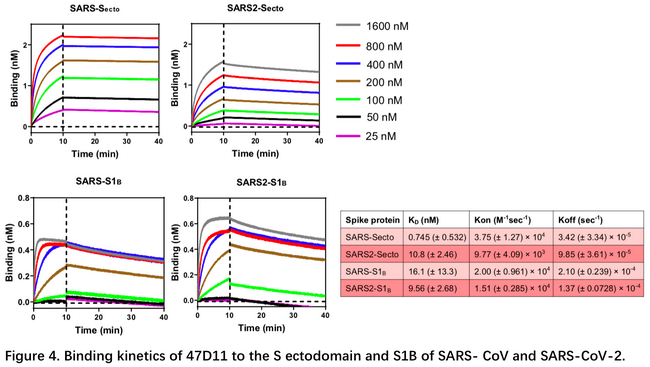
3月11日荷兰乌得勒支大学的研究者在bioRxiv发表了首个全人源中和抗体4。文中为了得到一株可能的冠状病毒的广谱抗体,利用HCoV-OC43,SARS-CoV,MERS三个hCOV的S蛋白免疫H2L2小鼠,用ELISA检测得到4株对SARS-CoV S蛋白胞外域(Secto)和SARS-CoV-2 S1亚基均结合的单抗47D11,并将47D11重组表达为完全人 IgG1 同种型抗体。研究人员通过细胞实验确定47D11可中和SARS-CoV / SARS-CoV-2假病毒和真病毒侵染,然后利用BLI技术通过固定抗体,结合抗原,测定了47D11与SARS-CoV /SARS-CoV-2的Secto或S1b的结合解离。发现47D11对SARS-CoV-Secto的亲和力比SARS-CoV-2-Secto高,但对两种冠状病毒S1b的亲和力接近,与ELISA的结果吻合。后续通过流式细胞分选实验发现47D11和ACE2对S蛋白的表位并不一致,而在细胞融合实验中47D11可以减少SARS-CoV和SARS-CoV-2的S蛋白介导的合胞体形成,以上说明47D11可能是结合在S蛋白更保守的位置,因此赋予了它潜在的广谱抑制能力。
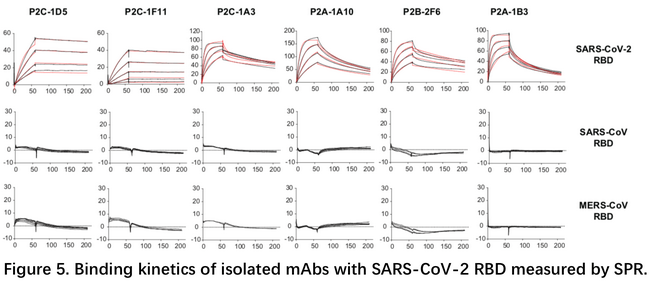
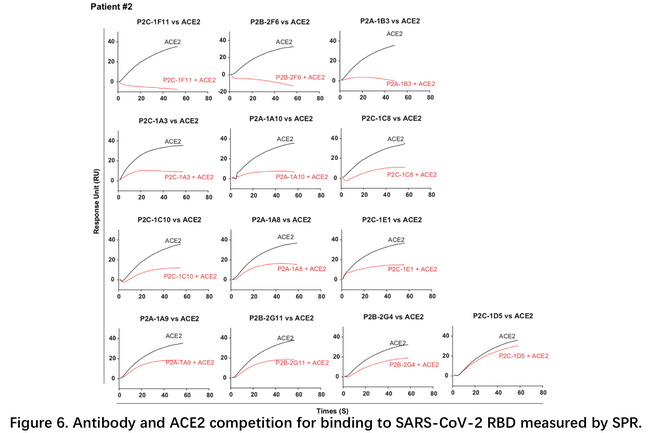
在与清华大学王新泉团队合作发表了复合物结构后,3月21日清华大学张林琦团队与深圳三院合作发表了研究成果,团队从患者血液B淋巴细胞中,成功分离出206株抗SARS-CoV-2的单克隆抗体,其中一些在假病毒和活病毒中和实验中表现良好5。文中做了大量的SPR实验来分别检测了抗体与RBD的结合解离,抗体与ACE2竞争性结合RBD,抗体之间的表位分析。所测试的抗体多数对SARS和MERS的RBD没有交叉结合能力,有部分抗体与ACE2完全竞争性结合RBD,如P2B-2F6。抗体之间的表位分析发现之间的竞争关系差异很大,如P2C-1A3几乎与其他所有的抗体都竞争,而P2C-1F11则相反,仅与P2C-1C10有微弱的竞争。这说明不同的抗体其中和机制不尽相同。

抑制剂
针对病毒入侵细胞的过程,MIT的研究团队根据ACE2与RBD复合物的结构构建出ACE2的一段多肽,发现其可能有潜在的抑制病毒入侵能力,这一结果在3月20日发表在bioRxiv6。文章首先合成了ACE2肽酶结构域的一段a-helix(23AA) SBP1,利用BLI技术发现SBP1和RBD的KD达到47nM,与ACE2/RBD的亲和力比较接近,而更短的SBP2则失去了结合能力。研究团队推测SBP1可能会封闭ACE2与RBD的结合,抑制病毒入侵。

新的小分子药物开发周期较长,研究者们希望从已有的小分子药物中找到SARS-CoV-2的抑制剂。华东理工大学李洪林团队和武汉大学徐可团队3月13日合作在bioRxiv发文,发现靶向宿主细胞DHODH(二氢乳清酸脱氢酶,嘧啶合成途径中的关键酶)的抑制剂S312和S416有良好的广谱抗RNA病毒效果7。相较于靶向病毒分子的药物(DAA),靶向宿主细胞的药物(HTA)更能经受病毒变异的考验,有潜在的广谱抗病毒效果。李洪林团队前期通过小分子筛选,复合物结构分析,药物设计等多轮实验得到新型强效候选药物S312和S416,联合徐可团队在COVID-19疫情期间开展了两种候选药物对SARS-CoV-2的研究。

作者首先利用ITC测定了两种抑制剂的亲和力和自由能,又利用SPR测定了亲和力和结合解离速率。S312/S416与DHODH的KD值分别为20.3nM和1.69nM,并且呈现出快结合慢解离的特点。团队在细胞水平测试了S312/S416/Leflunomide/Teriflunomide(已上市的DHODH抑制剂药物,适应症为炎症等)对几种流感病毒,Ebola,Zika,SARS-CoV-2的抑制效果,发现抑制能力多为S416 > S312 > Teriflunomide> Leflunomide,其中S416对SARS-CoV-2的EC50为17nM,SI>5882。在流感病毒小鼠实验中,团队发现DAA和HTA联合用药更有效。而作为DHODH抑制剂,S312/S416同时可以抑制免疫细胞,这可能有助于病人的临床治疗。
疫苗
分子间相互作用技术除了检测分子间的相互作用,还可以定量检测抗体,适用于疫苗免疫后抗体产生水平的测定,在重组蛋白疫苗的研发中还可用于抗原的检测。在疫苗生产中也可以作为质控的手段。
总结
除了得到KD外,分子间相互作用技术中ITC可以得到更多的热力学信息,如焓变,熵变;BLI和SPR通过实时测定结合和解离过程,得到更多结合解离信息,同时能直观,快速地做抗体表位分析;MST实验中,其热场中分子的泳动行为不仅和分子大小有关还与水化层及表面电荷有关;AUC得到的样品各组分的信息里,还能分析得到分子间的结合比例。
蛋白质制备与鉴定平台提供多种分子间相互作用技术的开放服务,可适用小分子,蛋白,核酸,多糖,纳米颗粒,细菌等各类型样品,平台同时有多种生物分子的性质鉴定仪器,包括分析超速离心AUC,圆二色光谱仪CD,差示量热扫描仪DCS,动态光散射仪DLS,十八度角静态光散射仪SLS等等。
蛋白质制备与鉴定平台链接:http://phoenix.tsinghua.edu.cn/index.php?c=show&id=85
文中若有错误之处还请批评指正。
参考文献:
1. Zhou, P., Yang, X., Wang, X. et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature 579, 270–273 (2020). https://doi.org/10.1038/s41586-020-2012-7
2. DANIEL W., NIANSHUANG W., KIZZMEKIA S. et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. SCIENCE13 MAR 2020 : 1260-1263
3. Xiaolong Tian, Cheng Li, Ailing Huang, et al. (2020) Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody, Emerging Microbes & Infections, 9:1, 382-385 DOI: 10.1080/22221751.2020.1729069
4.Chunyan W., Wentao L., Dubravka D., et al. A human monoclonal antibody blocking SARS-CoV-2 infection bioRxiv 2020.03.11.987958; doi: https://doi.org/10.1101/2020.03.11.987958
5. Bin J., Qi Z., Xiangyang G., et al. Potent human neutralizing antibodies elicited by SARS-CoV-2 infection bioRxiv 2020.03.21.990770; doi: https://doi.org/10.1101/2020.03.21.990770
6. G. Zhang, S. Pomplun, A. R, et al. The first-in-class peptide binder to the SARS-CoV-2 spike protein bioRxiv 2020.03.19.999318; doi: https://doi.org/10.1101/2020.03.19.999318
7. Rui X., Leike Z., Shiliang L., et al. Novel and potent inhibitors targeting DHODH, a rate-limiting enzyme in de novo pyrimidine biosynthesis, are broad-spectrum antiviral against RNA viruses including newly emerged coronavirus SARS-CoV-2 bioRxiv 2020.03.11.983056; doi: https://doi.org/10.1101/2020.03.11.983056
本文章版权归清华大学蛋白质研究技术中心蛋白质制备与鉴定平台所有





